Hélice alpha
L’hélice alpha (hélice α) est une structure secondaire courante des protéines. Elle est formée par une chaîne polypeptidique de forme hélicoïdale à pas de rotation droit dans laquelle chaque groupe N-H de la chaîne principale d'un acide aminé forme une liaison hydrogène avec le groupe C=O de la chaîne principale du quatrième acide aminé le précédant. Cette structure secondaire est parfois appelée hélice α de Pauling-Corey-Branson. Le nom d’hélice 3,613 est également utilisé, la valeur de 3,6 correspondant au nombre d'acides aminés dans le pas de l'hélice et 13 est le nombre d'atomes dans le cycle fermé par la liaison hydrogène.

Parmi les types de structures locales présents dans les protéines, l'hélice α est la structure secondaire la plus prévisible par l'analyse de la séquence primaire, ainsi que la plus répandue.
Découverte
modifierDans les années 1930, William Astbury met en évidence des changements très importants par diffraction de rayon X de fibres de laine ou de cheveux humides lors de leur étirement. Les données montrent que les fibres non étirées présentent une structure moléculaire répétitive tous les 5,1 Å (0,51 nm).
Astbury émet l'hypothèse que les fibres non étirées forment une structure repliée. Plus tard avec d'autres chercheurs notamment Maurice Huggins, il propose le modèle suivant :
- les molécules de protéines repliées forment une hélice appelée forme α ;
- l'étirement de la fibre provoque le déroulement de l'hélice formant un état étendu appelé forme β.
Hans Neurath (en) est le premier à mettre en question une partie des modèles d'Astbury pour des problèmes d'effets stériques entre atomes[1]. Bien que le modèle d'Astbury soit incorrect, il est initiateur des modèles plus récents de structure secondaire pour les protéines avec l'hélice α et le feuillet β (la nomenclature de Astbury a été conservée), développés par Linus Pauling, Robert Corey et Herman Branson en 1951 et proposent l'existence d'hélice droite et d'hélice gauche sur des considérations théoriques. En 1960, la structure cristalline de la myoglobine montre la présence majoritaire d'hélices droites[2]. Les travaux de Neurath et d'Astbury ont permis à HS Taylor (en)[3], Maurice Huggins (en)[4] et William Bragg[5] de proposer un modèle proche de l'hélice α pour la kératine.
Deux éléments cruciaux ont permis l'établissement des caractéristiques de l'hélice α : d'une part la connaissance de la géométrie des liaisons obtenue grâce aux structures cristallographiques de peptides et la prédiction de Pauling sur le caractère plan de la liaison peptidique et d'autre part l'abandon de l'hypothèse d'un nombre entier d'acides aminés par tour d'hélice. Le moment clé de la découverte du modèle de l'hélice α se déroule au cours du printemps 1948. Durant cette période Pauling tombe malade et doit rester chez lui : désœuvré, il dessine une chaîne de polypeptides sur une bande de papier en veillant à conserver les liaisons peptidiques planes. Après quelques tentatives, il parvient à obtenir un modèle avec des liaisons hydrogène physiquement plausibles. Pauling travaille ensuite avec Corey et Branson pour confirmer son modèle avant publication[6]. En 1954, Pauling reçoit son premier prix Nobel pour ses recherches sur la nature de la liaison chimique et son application à l'élucidation de la structure des substances complexes[7], telles que les protéines et la structure de l'hélice α.
Structure
modifierGéométrie et liaisons hydrogène
modifier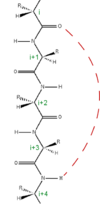
Les acides aminés dans une hélice α sont organisés dans une structure hélicoïdale avec un pas de rotation droit où chaque acide aminé forme un angle de 100° dans l'hélice et entraine une translation 1,5 Å (0,15 nm) le long de l'axe de l'hélice α. Dunitz[8] décrit comment dans le premier article de Pauling sur le thème ce dernier décrit une hélice gauche, énantiomère de la véritable structure. Il existe cependant quelques courtes hélices à pas de rotation gauche composées majoritairement de glycine, le seul acide aminé achiral, les autres acides aminés biologiques chiraux L déstabilisent ce type de structure. Le pas de l'hélice α (la distance verticale entre deux spires consécutives de l'hélice) est de 5,4 Å (0,54 nm), correspondant au produit de 1,5 et 3,6. L'élément le plus caractéristique de l'hélice α est la liaison hydrogène entre le groupe N-H de la chaine principale d'un acide aminé et le groupe C=O de la chaine principale du quatrième acide aminé le précédant. La nomenclature officielle internationale[9],[10] propose deux façons de définir une hélice α, la règle 6.2 décrit la répétition des angles de torsions φ, ψ des acides aminés impliqués dans l'hélice α ; la règle 6.3 décrit l'hélice α comme un motif géométrique caractérisé par la hauteur du pas et les liaisons hydrogène. Les hélices α peuvent être identifiées et prédites dans les structures de protéines à l'aide de méthode de calcul de prédiction dont l'une est le DSSP (Dictionnaire de Structures Secondaires des Protéines)[11].
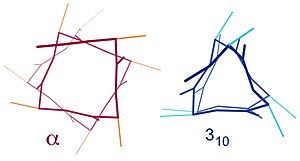
Des structures en hélice similaires comprennent les hélices 310 (présentant une liaison hydrogène sur la chaîne principale tous les trois acides aminés) et les hélices π (présentant une liaison hydrogène tous les cinq acides aminés). L'hélice α peut être décrite comme une hélice 3,613, dans cette représentation, la valeur en indice (13 pour l'hélice α) indique le nombre d'atomes impliqués dans la boucle formée par la liaison hydrogène ; le nombre 3,6 indique le nombre d'acides aminés par tour d'hélice[12].

La chaine principale des acides aminés impliqués dans une hélice α possède des angles dièdres (φ, ψ) dont les valeurs moyennes sont (-60°, -45°) comme indiqué dans le diagramme de Ramachandran ci-contre. Plus généralement, la somme de l'angle dièdre ψ d'un acide aminé et de l'angle dièdre φ de l'acide aminé suivant est proche de -105° ; en conséquence les angles dièdres des acides aminés formant une hélice α forme une diagonale sur le diagramme de Ramachandran entre les valeurs (-90°, -15°) et (-35°, -70°). Pour comparaison la somme des angles dièdres pour les hélices 310 est de -75°, tandis qu'il est proche de -130° pour les hélices-π. La formule générale de l'angle de rotation Ω par acide aminé dans les différents types d'hélice polypeptidique formés d'isomères trans est donnée par l'équation[14] :
L'hélice α a une structure particulièrement compacte, il n'existe pratiquement aucun espace libre à l'intérieur de l'hélice. Les chaines latérales des acides aminés sont situées à l'extérieur de l'hélice et sont légèrement orientées dans la direction de l'extrémité N-terminale (cette orientation est parfois comparée à l'organisation des branches de sapins de Noël autour du tronc). Cette propriété peut être utilisée en basse résolution sur des cartes de densité électronique pour déterminer le sens du squelette carboné de la protéine étudiée[15].

Diagramme 2D de représentation d'hélice α
modifier
Trois formes de diagramme 2D de représentation d'hélice α sont utilisées pour présenter les relations entre la structure primaire de la chaine peptidique et la structure secondaire. Il est alors possible de visualiser les propriétés physiques et les interactions spécifiques des différentes chaines latérales des acides aminés impliqués dans l'hélice α. Deux d'entre elles permettent de modéliser l'hélice α autour de la section cylindrique : la représentation en roue hélicoïdale[16] (Helical Wheel) et plus récemment la représentation de Wenxiang (en) (Wenxiang diagram)[17].
La représentation en roue hélicoïdale modélise l'hélice α par projection du squelette carboné de la chaine principale des Cα des acides aminés autour de l'axe de l'hélice du sommet vers le bas (voir le modèle ci-contre). La représentation de Wenxiang est plus abstraite en forme de spirale enroulée sur le plan de la feuille. Les deux représentations permettent la visualisation de la séquence d'acides aminés en utilisant le codage à une lettre sur chaque position du Cα, en utilisant des couleurs ou des symboles particuliers pour décrire les propriétés des acides aminés. Les acides aminés hydrophobes et hydrophiles sont toujours signalés, ils sont en effet impliqués dans les interactions de l'hélice α avec environnement proche. Dans certaines représentations les acides aminés chargés positivement et négativement sont indiqués ainsi que les acides aminés ambigus comme la glycine. Il n'existe pas de convention standardisée pour le codage des chaines latérales d'acides aminés dans ces modèles. La représentation de Wenxiang permet de visualiser les positions relatives des acides aminés les uns par aux autres pour des hélices α de toute longueur alors la représentation en roue hélicoïdale est limitée à des hélices α courtes du fait de la superposition des acides aminés dans le modèle.
Ces différents modèles apportent une représentation intuitive et visuelle en 2D de la disposition des acides aminés hydrophobes et hydrophiles sur l'hélice α[16],[17], ils peuvent être utilisés pour étudier les interactions hélice/hélice[18], hélice/membranes[19] ou protéine/protéine[20],[21]. Plusieurs sites internet et programmes sont disponibles pour générer des représentations en roue hélicoïdale[22], plus récemment un serveur web appelé Wenxiang[23] a été créé pour permettre de tracer la représentation de Wenxiang de toute hélice α[24].
La dernière représentation 2D de l'hélice α consiste à ouvrir l'hélice le long d'une ligne parallèle à l'axe de l'hélice, puis de représenter les acides aminés verticalement. Cette représentation n'est pas adaptée aux interactions entre hélices mais c'est un moyen de visualiser l'arrangement de séquence pour les protéines membranaires intrinsèques en montrant le positionnement des acides aminés hydrophobes en interaction avec la membrane sans la nécessité de connaitre la disposition dans l'espace des hélices α.
Stabilité
modifierLes hélices α étudiées dans les protéines sont formées de quatre à plus de quarante résidus, une hélice α moyenne contient environ dix acides aminés et forme environ trois tours. En général, les polypeptides courts ne présentent pas de structure en hélice α en solution, le coût entropique du repliement de la chaine polypeptidique n'est pas compensé par une quantité suffisante d'interactions stabilisantes. Les liaisons hydrogène présentes au sein des hélices α sont considérées comme plus faibles que celles trouvées dans les feuillets β et sont facilement attaquées par les molécules d'eau. Dans des environnements hydrophobes comme dans la membrane plasmique, en présence de mélange de solvant eau trifluoroéthanol (TFE) ou isolés en phase gazeuse[25], les polypeptides peuvent adopter des structures stables en hélice α. L'ajout de molécules dites crosslinks dans des solutions de peptides peut stabiliser la structure en hélice.

Techniques expérimentales identifiant les hélices α
modifierDepuis la caractérisation des hélices α par leurs liaisons hydrogène et le squelette carboné de la chaine principale des acides aminés, la preuve expérimentale la plus détaillée de la structure de l'hélice α provient des analyses obtenues par cristallographie aux rayons X (comme dans l'exemple ci-contre). Les atomes d'oxygène des groupes carbonyle du squelette peptidique pointent vers le bas (en direction du C-terminale), mais s'évasent légèrement ; les liaisons hydrogène sont pratiquement parallèles à l'axe de l'hélice. Les études structurales de protéines réalisées par spectroscopie RMN des protéines (en) décrivent de façon similaire les hélices α grâce à l'étude des effets Overhauser nucléaire (effet NOE) montrant des couplages entre atomes sur spires adjacentes. Dans certaines cas, les liaisons hydrogène individuelles peuvent être identifiées comme un faible couplage en spectroscopie RMN.
Il existe plusieurs méthodes à basse résolution permettant de caractériser l'hélice α. Le déplacement chimique en spectroscopie RMN des carbones Cα, Cβ, C' ainsi que les couplages dipolaires résiduels (en) sont suffisants pour la caractérisation. L'étude du dichroïsme circulaire d'hélices α dans les UV-lointains (170-250 nm) est caractéristique, deux minimums sont observés vers 208 nm et 222 nm. La caractérisation des hélices α par spectroscopie infrarouge est rarement utilisée car le spectre des hélices α présente trop de similitudes avec celui des structures aléatoires, il est cependant possible de discriminer les deux types de structure par un échange hydrogène-deutérium (en). La microscopie électronique à basse température permet maintenant d'identifier individuellement les hélices α au sein d'une protéine, la caractérisation des différents acides aminés formant l'hélice est un domaine de recherche actif.
Les longues chaines d'homopolymères d'acides aminés forment souvent, lorsqu'elles sont solubles, des hélices α. Ces longues hélices peuvent être détectées par relaxation diélectrique, par biréfringence d'écoulement (en) ou par mesure de constante de diffusion.
Acides aminés impliqués
modifierPlusieurs acides aminés favorisent la formation d'hélices α comme la méthionine, l'alanine, la leucine, le glutamate non chargé ou la lysine (ou « MALEK » en utilisant la notation des acides aminés à une lettre). En revanche, la proline et la glycine ont un rôle déstabilisant dans la création des hélices α[26]. La proline ne possède pas d'hydrogène sur sa chaine principale et ne peut pas former de liaison hydrogène, de plus sa chaine latérale interfère stériquement avec le squelette carboné du tour précédent. Si une proline était présente au sein d'une hélice α, elle provoquerait l'arrêt de l'hélice ou une forte déformation d'environ 30° par rapport à l'axe de l'hélice[12]. En revanche la proline est souvent le premier acide aminé d'une hélice α probablement du fait de sa rigidité structurelle. La présence de glycine dans une hélice α a tendance à perturber la structure du fait de la grande flexibilité conformationnelle de cet acide aminé.
Tableau d'affinité des acides aminés dans la formation d'hélices α
modifierLe tableau présente une moyenne de la différence d'énergie libre Δ(ΔG) exprimée en Kcal/mol de chaque acide aminé dans une hélice α par rapport à l'énergie libre de l'alanine (arbitrairement fixée à 0). Les nombres positifs les plus élevés indiquent les acides aminés les moins favorables pour former une hélice α. Des écarts dans ces valeurs peuvent cependant être observés selon la nature des résidus voisins.
| Acide aminé | Code 3 lettres | Code 1 lettre | Δ(ΔG) (kcal/mol (en)[26]) |
|---|---|---|---|
| Alanine | Ala | A | 0 |
| Arginine | Arg | R | 0,21 |
| Asparagine | Asn | N | 0,65 |
| Acide aspartique | Asp | D | 0,69 |
| Cystéine | Cys | C | 0,68 |
| Acide glutamique | Glu | E | 0,40 |
| Glutamine | Gln | Q | 0,39 |
| Glycine | Gly | G | 1,00 |
| Histidine | His | H | 0,61 |
| Isoleucine | Ile | I | 0,41 |
| Leucine | Leu | L | 0,21 |
| Lysine | Lys | K | 0,26 |
| Méthionine | Met | M | 0,24 |
| Phénylalanine | Phe | F | 0,54 |
| Proline | Pro | P | 3,16 |
| Sérine | Ser | S | 0,50 |
| Thréonine | Thr | T | 0,66 |
| Tryptophane | Trp | W | 0,49 |
| Tyrosine | Tyr | Y | 0,53 |
| Valine | Val | V | 0,61 |
Moment dipolaire
modifierUne hélice α dispose d'un moment dipolaire global correspondant à la somme des micro-dipôles des groupes carbonyle le long de l'axe de l'hélice[27]. Les effets de ce moment dipolaire au niveau de l'hélice est sujet à controverses. L'extrémité N-terminale des hélices α est souvent associée à une charge négative provenant d'une chaine latérale d'un acide aminé (glutamate ou aspartate) ou parfois d'un groupe phosphate. Plusieurs scientifiques considèrent que le moment dipolaire de l'hélice est associé à cette interaction électrostatique, tandis que d'autres que le groupe NH libre à l'extrémité N-terminale pourrait être modélisé comme une liaison hydrogène[28],[29].
Superhélice « Coiled coil »
modifierLes superhélices (coiled coil) sont des formes très stables dans lesquelles au moins deux hélices α s'enroulent les unes aux autres dans une super structure secondaire. Les hélices composant cette structure sont formées d'une succession de motifs périodiques de sept résidus. Le premier et le quatrième acide aminé (connus sous leurs positions a et d) sont pratiquement toujours hydrophobes (le résidu d est souvent une leucine) sont orientés vers l'intérieur du motif en superhélice. En général, le cinquième et septième résidu (position e et g) sont des acides aminés de charge opposée et forment un pont salin stabilisé par des interactions électrostatiques. Les protéines fibreuses comme la kératine ou les tiges de la myosine ou de la kinésine adoptent souvent des structures de superhélice comme les protéines dimérisées. Une paire de superhélices (formant un faisceau de quatre hélices) est une structure très commune chez les protéines, on le retrouve dans l'hormone de croissance humaine et dans plusieurs variétés de cytochromes. La protéine Rop, favorisant la réplication de plasmide dans les bactéries est un cas unique ou la chaine polypeptidique forme une superhélice et l'association de deux monomères s'assemblent pour former un faisceau à quatre hélices.
Protéines α
modifier
La myoglobine et l'hémoglobine sont les premières protéines dont les protéines ont été résolues par cristallographie aux rayons X ; elles ont des repliements très proches et sont formées à plus de 70% d'hélices α, le reste des acides aminés forment des boucles sans structure secondaire caractéristique. La classification structurale des protéines basée sur les domaines structuraux dispose d'une base de données composée de la plupart des types de protéines α.
Rôle fonctionnel
modifier

Interactions avec l'ADN
modifierDes hélices α sont présentes dans les motifs structuraux de liaison avec l'ADN, comme les motifs hélice-coude-hélice, glissière à leucine et doigt de zinc. Le diamètre d'une hélice α avec des chaines latérales d'acides aminés est d'environ 12 Å (soit 1,2 nm) correspondant à la longueur du grand sillon d'une molécule d'ADN de forme B. Les motifs structuraux en superhélice ou en glissière sont des dimères d'hélices qui peuvent interagir de façon symétrique de part et d'autre de la molécule d'ADN. Le facteur de transcription Max présente une glissière à leucine composée d'une superhélice organisée en dimère portant deux autres hélices interagissant avec deux spires consécutives du grand sillon de l'ADN.
Protéines membranaires
modifierLes hélices α sont des composants classiques de la structure des protéines transmembranaires, les liaisons hydrogène formant la structure de l'hélice sont enfouies dans le corps de la structure secondaire, les chaines latérales hydrophobes d'acides aminés interagissent alors avec les membranes. Les protéines transmembranaires sont ancrées dans la membrane par un nombre variable d'hélice α, une des structures les plus communes est formée de sept hélices transmembranaires formant un anneau comme dans le cas de la rhodopsine bovine ou du récepteur couplé aux protéines G (RCPG).
Propriétés mécaniques
modifierLes hélices α peuvent subir des contraintes d'étirement suivant l'axe de l'hélice, observées sur des filaments ou des tissus riche en hélices α. Cet étirement est caractérisé par trois phases[30] : la phase I correspond à un régime de faibles déformations au cours duquel l'hélice est étirée de manière homogène ; la phase II correspond à la désorganisation des spires de l'hélice par des ruptures de liaisons hydrogène enfin la phase III est associée aux déformations des liaisons covalentes dans l'hélice.
Caractéristiques dynamiques
modifierLes hélices α dans les protéines peuvent présenter un mouvement à basse fréquence en accordéon observé par spectroscopie Raman[31] et caractérisé par le modèle de quasi-continuum[32],[33]. Les hélices non stabilisées par des interactions de structures tertiaires présentent un comportement dynamique principalement attribué à une dégradation progressive de l'hélice à ses extrémités[34].
Transition hélice - bobine
modifier
Des homopolymères d'acides aminés comme les poly-lysines sont susceptibles d'adopter une structure d'hélice α à basse température se dégradant à haute température. Cette transition hélice - bobine a été souvent considérée comme analogue à la dénaturation des protéines. Il est possible de modéliser cette transition par physique statistique en utilisant une matrice de transfert basée sur deux paramètres, la propension à initier une hélice et la propension à étirer une hélice.
L'hélice α dans l'art
modifierAu moins cinq artistes ont présenté des références à l'hélice α dans leur travail : Julie Newdoll en peinture, Julian Voss-Andreae, Bathsheba Grossman, Byron Rubin, et Mike Tyka en sculpture.
Julie Newdoll détient un diplôme en microbiologie avec une mineure en art, s'est spécialisée dans des peintures inspirées d'images et de molécules microscopiques depuis 1990. Sa peinture Rise of the Alpha Helix (en 2003) présente des figures humaines disposées dans un agencement d'hélice α.
Julian Voss-Andreae est un sculpteur d'origine allemande diplômé en physique expérimentale et en sculpture. Depuis 2001, il réalise des sculptures ayant pour modèle les structures des protéines[36], l'hélice α étant l'un de ses sujets préférés. Il a réalisé des sculptures d'hélice α avec différents matériaux, notamment du bambou ou des arbres entiers. En 2004, il a réalisé un monument pour honorer la mémoire de Linus Pauling, l'homme qui a découvert l'hélice α. Cette sculpture est faite d'une grande poutre en acier qui a été modifiée de façon à recréer la structure de l'hélice α. Cette sculpture rouge vif de 3 m de hauteur est installée devant la maison où Pauling a passé son enfance, à Portland dans l'Oregon.
Notes et références
modifier- (en) H Neurath, « Intramolecular folding of polypeptide chains in relation to protein structure », Journal of Physical Chemistry, vol. 44, no 3, , p. 296-305 (DOI 10.1021/j150399a003)
- (en) JC Kendrew, RE Dickerson, BE Strandberg, RG Hart, DR Davies, DC Phillips et VC Shore, « Structure of myoglobin : A three-dimensional Fourier synthesis at 2 Å resolution », Nature, vol. 185, no 4711, , p. 422-427 (PMID 18990802, DOI 10.1038/185422a0).
- (en) HS Taylor, « Large molecules through atomic spectacles », Proceedings of the American Philosophical Society, vol. 85, , p. 1-12.
- (en) M Huggins, « The structure of fibrous proteins », Chemical Reviews, vol. 32, no 2, , p. 195-218 (DOI 10.1021/cr60102a002).
- (en) WL Bragg, JC Kendrew et MF Perutz, « Polypeptide chain configurations in crystalline proteins », Proceedings of the Royal Society A, vol. 203, no 1074, , p. 321-357 (DOI 10.1098/rspa.1950.0142, lire en ligne).
- (en) L Pauling, RB Corey et HR Branson, « The Structure of Proteins : Two Hydrogen-Bonded Helical Configurations of the Polypeptide Chain », Proceedings of the National Academy of Sciences of the United States of America, vol. 37, no 4, , p. 205-211 (PMID 14816373, PMCID 1063337, DOI 10.1073/pnas.37.4.205).
- lire en ligne
- (en) J Dunitz, « Pauling's Left-Handed α-Helix », Angewandte Chemie International Edition, vol. 40, no 22, , p. 4167-4173.
- (en) IUPAC-IUB Commission on Biochemical Nomenclature, « Abbreviations and symbols for the description of the conformation of polypeptide chains », Journal of Biological Chemistry, vol. 245, , p. 6489–6497 (lire en ligne).
- lire en ligne.
- K Kabsch et C Sander, « Dictionary of protein secondary structure : pattern recognition of hydrogen-bonded and geometrical features », Biopolymers, vol. 22, no 12, , p. 2577–2637 (PMID 6667333, DOI 10.1002/bip.360221211).
- JS Richardson, « The Anatomy and Taxonomy of Proteins », Advances in Protein Chemistry, vol. 34, , p. 167-339[1] (PMID 7020376, DOI 10.1016/S0065-3233(08)60520-3).
- (en) SC Lovell, IW Davis, VW III Arendall, PIW de Bakker, JM Word, MG Prisant, JS Richardson et DC Richardson, « Structure validation by Cα geometry : φ,ψ and Cβ deviation », Proteins, vol. 50, no 3, , p. 437–450 (PMID 12557186, DOI 10.1002/prot.10286).
- (en) RE Dickerson et I Geis, Structure and Action of Proteins, Harper, New York,
- (en) TC Terwilliger, « Rapid model-building of α-helices in electron density maps », Acta Crystallographica, vol. D66, , p. 268–275 (PMID 20179338, PMCID 2827347, DOI 10.1107/S0907444910000314).
- (en) M Schiffer et AB Edmundson, « Use of helical wheels to represent the structures of proteins and to identify segments with helical potential », Biophys. J., vol. 7, no 2, , p. 121–35 (PMID 6048867, PMCID 1368002, DOI 10.1016/S0006-3495(67)86579-2, Bibcode 1967BpJ.....7..121S)
- (en) KC Chou, CT Zhang et GM Maggiora, « Disposition of amphiphilic helices in heteropolar environments », Proteins, vol. 28, no 1, , p. 99–108 (PMID 9144795, DOI 10.1002/(SICI)1097-0134(199705)28:1<99::AID-PROT10>3.0.CO;2-C)
- (en) N Kurochkina, « Helix-helix interactions and their impact on protein motifs and assemblies », J. Theor. Biol., vol. 264, no 2, , p. 585–92 (PMID 20202472, DOI 10.1016/j.jtbi.2010.02.026)
- (en) D Eisenberg, RM Weiss et TC Terwilliger, « The helical hydrophobic moment : a measure of the amphiphilicity of a helix », Nature, vol. 299, no 5881, , p. 371–374 (PMID 7110359, DOI 10.1038/299371a0)
- (en) GP Zhou, « The disposition of the LZCC protein residues in wenxiang diagram provides new insights into the protein-protein interaction mechanism », J. Theor. Biol., vol. 284, no 1, , p. 142–148 (PMID 21718705, DOI 10.1016/j.jtbi.2011.06.006)
- (en) GP Zhou, « The Structural Determinations of the Leucine Zipper Coiled-Coil Domains of the cGMP-Dependent Protein Kinase I alpha and its Interaction with the Myosin Binding Subunit of the Myosin Light Chains Phosphase », Protein & Peptide Letters, vol. 18, no 10, , p. 966-978 (PMID 21592084, DOI 10.2174/0929866511107010966)
- lire en ligne
- lire en ligne
- (en) KC Chou, WZ Ling et X Xiai, « Wenxiang : a web-server for drawing wenxiang diagrams », Natural Science, vol. 3, no 10, , p. 862-865 (DOI 10.4236/ns.2011.310111)
- (en) RR Hudgins et MF Jarrold, « Helix Formation in Unsolvated Alanine-Based Peptides : Helical Monomers and Helical Dimers », Journal of the American Chemical Society, vol. 121, no 14, , p. 3494-3501 (DOI 10.1021/ja983996a)
- (en) CN Pace et JM Scholtz, « A helix propensity scale based on experimental studies of peptides and proteins », Biophys. J., vol. 75, no 1, , p. 422–427 (PMID 9649402, PMCID 1299714, DOI 10.1016/S0006-3495(98)77529-0, Bibcode 1998BpJ....75..422N)
- (en) WGJ Hol et PT van Duijnen, « The alpha helix dipole and the properties of proteins », Nature, vol. 273, , p. 443–446 (DOI 10.1038/273443a0)
- (en) JJ He et FA Quiocho, « Dominant role of local dipoles in stabilising uncompensated charges in a sulfate sequestered in a periplasmic active transport protein », Protein Science, vol. 2, , p. 1643–1647 (DOI 10.1002/pro.5560021010)
- (en) EJ Milner-White, « The partial charge of the nitrogen atom in peptide bonds », Protein Science, vol. 6, , p. 2477–2482 (DOI 10.1002/pro.5560061125)
- (en) T Ackbarow, X Chen, S Keten et MJ Buehler, « Hierarchies, multiple energy barriers and robustness govern the fracture mechanics of alpha-helical and beta-sheet protein domains », Proceedings of the National Academy of Sciences of the United States of America, vol. 104, no 42, , p. 16410–16415 (PMID 17925444, PMCID 2034213, DOI 10.1073/pnas.0705759104)
- (en) PC Painter, LE Mosher et C Rhoads, « Low-frequency modes in the Raman spectra of proteins », Biopolymers, vol. 21, no 7, , p. 1469–1472 (PMID 7115900, DOI 10.1002/bip.360210715)
- (en) KC Chou, « Identification of low-frequency modes in protein molecules », Biochem. J., vol. 215, no 3, , p. 465–469 (PMID 6362659, PMCID 1152424)
- KC Chou, « Biological functions of low-frequency vibrations (phonons). III. Helical structures and microenvironment », Biophys. J., vol. 45, no 5, , p. 881–889 (PMID 6428481, PMCID 1434967, DOI 10.1016/S0006-3495(84)84234-4, Bibcode 1984BpJ....45..881C)
- (en) B Fierz, A Reiner et T Kiefhaber, « Local conformational dynamics in α-helices measured by fast triplet transfer », Proceedings of the National Academy of Sciences of the United States of America, vol. 106, no 44, , p. 1057-1062 (PMID 19131517, PMCID 2633579, DOI 10.1073/pnas.0808581106)
- La sculpture est installée devant la maison où Linus Pauling a passé son enfance, à Portland dans l’Oregon.
- (en) J Voss-Andreae, « Protein Sculptures: Life's Building Blocks Inspire Art », Leonardo, vol. 38, , p. 41–45 (DOI 10.1162/leon.2005.38.1.41)
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Alpha helix » (voir la liste des auteurs).
Voir aussi
modifierBibliographie
modifier- (en) John Tooze et Carl-Ivar Brändén, Introduction to protein structure, New York, Garland Pub, (ISBN 0-8153-2304-2).
- (en) D Eisenberg, « The discovery of the alpha-helix and beta-sheet, the principal structural features of proteins », Proceedings of the National Academy of Sciences of the United States of America, vol. 100, no 20, , p. 11207–11210 (PMID 12966187, PMCID 208735, DOI 10.1073/pnas.2034522100)
- (en) H Sugeta et T Miyazawa, « General Method for Calculating Helical Parameters of Polymer Chains from Bond Lengths, Bond Angles, and Internal-Rotation Angles », Biopolymers, vol. 5, no 7, , p. 673–679 (DOI 10.1002/bip.1967.360050708)
- (en) A Wada, « The alpha-helix as an electric macro-dipole », Advances in Biophysics, , p. 1–63 (PMID 797240)
- (en) WG Hol, « The role of the alpha-helix dipole in protein function and structure », Progress in Biophysics and Molecular Biology, vol. 45, no 3, , p. 149–195 (PMID 3892583, DOI 10.1016/0079-6107(85)90001-X)
- (en) DJ Barlow et JM Thornton, « Helix geometry in proteins », Journal of Molecular Biology, vol. 201, no 3, , p. 601–619 (PMID 3418712, DOI 10.1016/0022-2836(88)90641-9)
![{\displaystyle 3\cos \Omega =1-4\cos ^{2}\left[\left(\phi +\psi \right)/2\right]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/289a67463099a87877ac9fca00daf052bb2edd9a)