MNase-Seq
Le MNase-Seq est une technique utilisée en biologie moléculaire pour identifier les régions de la chromatine occupées par des nucléosomes à l'échelle du génome.
Description
modifier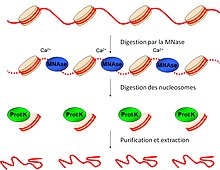
Cette technique repose sur le séquençage à haut débit de fragments issus de la digestion par MNase. La digestion de la chromatine par MNase est elle-même une méthode relativement simple et efficace qui permet d’identifier les régions d’ADN occupées par un ou plusieurs nucléosomes[1],[2]. Lorsque des noyaux de cellules perméabilisées sont exposés à la MNase, en présence d’un cation divalent (par exemple le Ca2+), cette nucléase effectue des coupures double-brin entre les nucléosomes, comme montré sur la figure ci-contre. Le traitement de substrats chromatiniens avec de très hautes concentrations en MNase produit une majorité de mononucléosomes. Une migration sur gel, suivie d’une coupure autour de 146 paires de base (p.b.) permet de s’assurer de l’obtention d’une grande majorité de mononucléosomes. Les fragments issus de ce processus sont ensuite séquencés selon les divers protocoles de séquençage à haut débit.
Traitement des données MNase-Seq
modifierLe traitement de base des données MNase-Seq est similaire à celui du ChIP-Seq, DNase-Seq et FAIRE-Seq, à l'exception près que les fragments sont en général étendus à exactement 146 p.b. de manière à refléter la taille réelle des fragments[3]. D'autres méthodes, basées sur un décalage des reads de 73 p.b. permettent également d'inférer le point médian nucléosomal, ou encore les extrémités des nucléosomes en n'effectuant aucune élongation pour chaque brin et en utilisant la coordonnée de départ de chaque read (orientée selon le brin)[4].
Articles connexes
modifierNotes et références
modifier- (en) R. J. Clark et G. Felsenfeld, « Structure of Chromatin », Nature New Biology, vol. 229, , p. 101–106 (DOI 10.1038/newbio229101a0, lire en ligne, consulté le )
- R. Axel, « Cleavage of DNA in nuclei and chromatin with staphylococcal nuclease », Biochemistry, vol. 14, , p. 2921–2925 (ISSN 0006-2960, PMID 1148185, lire en ligne, consulté le )
- Yong Zhang, Zarmik Moqtaderi, Barbara P. Rattner et Ghia Euskirchen, « Intrinsic histone-DNA interactions are not the major determinant of nucleosome positions in vivo », Nature Structural & Molecular Biology, vol. 16, , p. 847–852 (ISSN 1545-9985, PMID 19620965, PMCID 2823114, DOI 10.1038/nsmb.1636, lire en ligne, consulté le )
- Romain Fenouil, Pierre Cauchy, Frederic Koch et Nicolas Descostes, « CpG islands and GC content dictate nucleosome depletion in a transcription-independent manner at mammalian promoters », Genome Research, vol. 22, , p. 2399–2408 (ISSN 1549-5469, PMID 23100115, PMCID 3514669, DOI 10.1101/gr.138776.112, lire en ligne, consulté le )